Dans la vidéo du jour, on étudie l’hypothèse dite du « Monde à ARN », qui stipule que la vie sur Terre aurait, à un moment donné, reposé sur l’ARN, au lieu du binôme ADN/protéines. Voire que cette phase aurait pu avoir émergé spontanément d’un monde inerte, et constituer ainsi la première marche de l’apparition de la vie sur Terre
Comme à mon habitude, ci-dessous quelques compléments et précisions pour celles et ceux qui voudraient creuser
Le cas des virus
Les plus attentifs d’entre vous auront remarqué que j’ai savamment esquivé la question de savoir si les virus sont à considérer comme faisant partie ou non du vivant ! La question n’a pas de réponse unique, et dans cette vidéo j’ai plutôt choisi de me concentrer sur le couple ADN/protéines comme caractéristique de la vie sur Terre telle qu’on la connait.
Transcription et traduction
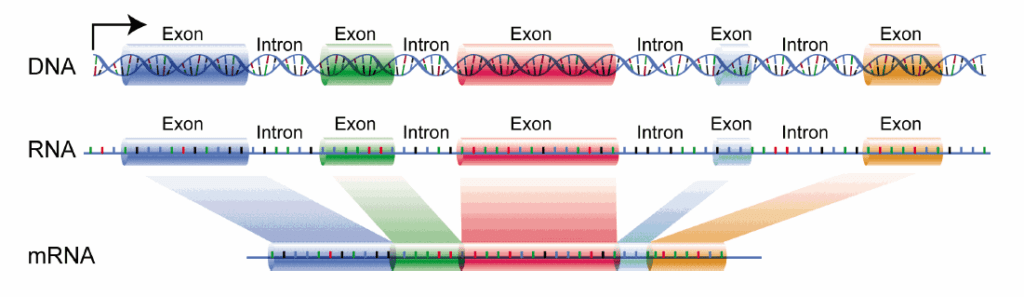
J’ai beaucoup simplifié tout ce qui tourne autour de l’information génétique et de sa circulation. Un point qui mérite d’être mentionné : tout l’ADN ne correspond pas à des gènes codant pour des protéines et donc tout l’ARN ne sera pas traduit. En effet notamment chez les eukaryotes, on distingue les introns (séquences non-codantes) et les exons (qui seront utilisés pour la traduction en protéines.)
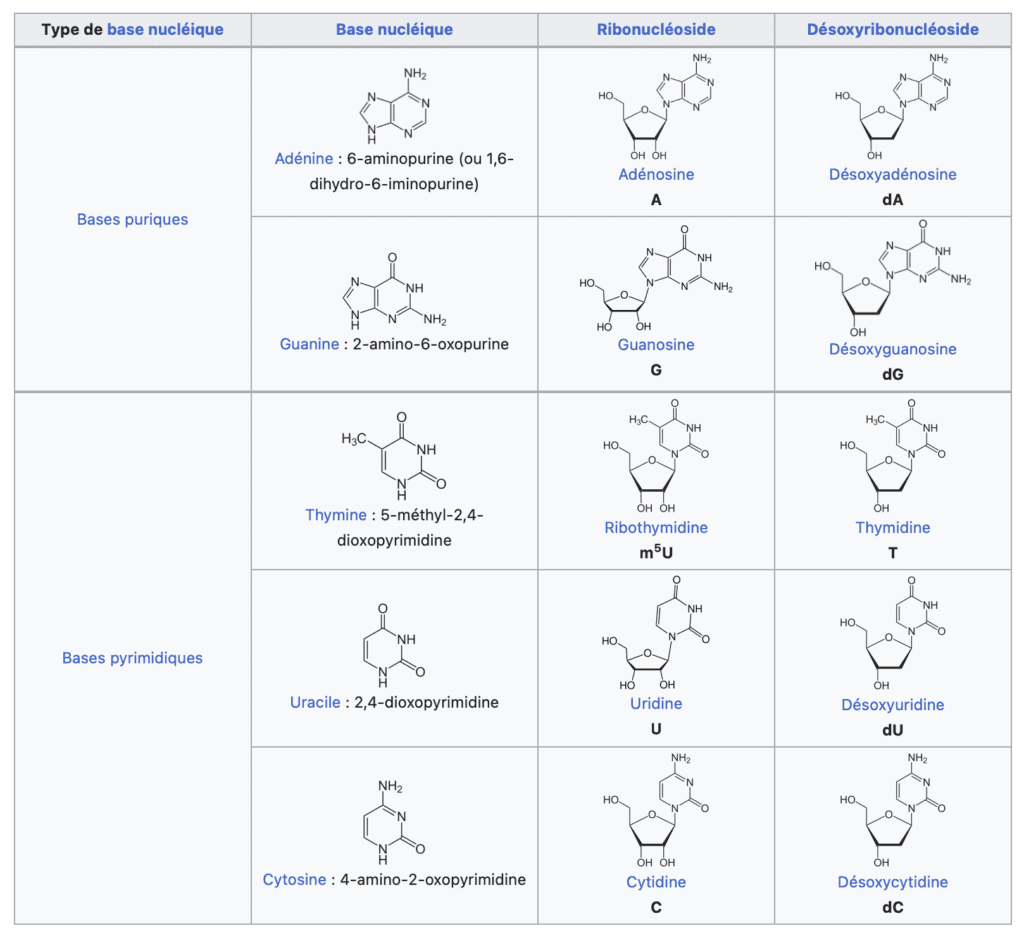
Nucléosides et nucléotides
J’ai balayé un peu vite le vocabulaire précis concernant la structure de l’ADN et de l’ARN. Techniquement on appelle nucléosides (avec un S) l’association d’une base et d’un ribose, alors que pour faire un nucléotide (avec T) il faut y ajouter le groupement phosphate qui va permettre d’une certaine façon la polymérisation.
Le tableau ci-dessous nous montre ça pour les différentes bases et les deux sucres
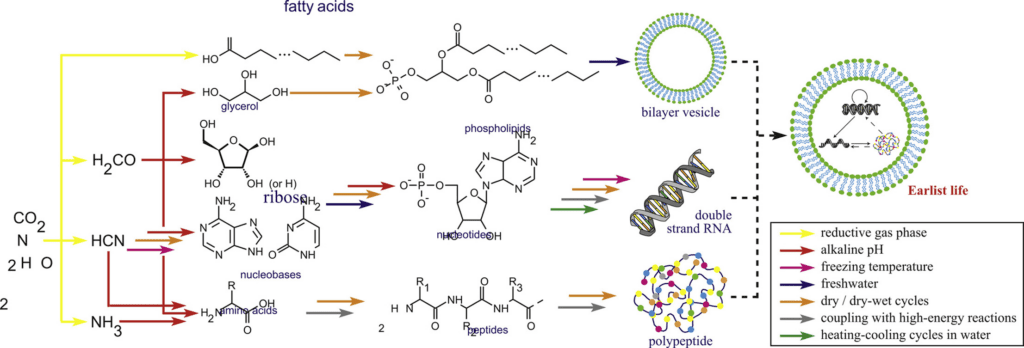
Et les compartiments ?
Dans les critères traditionnellement considérés pour définir la notion de système vivant, il y a l’idée d’une compartimentalisation. Par exemple avec une double membrane lipidique qui permet d’isoler les cellules vivantes de leur extérieur. Tout ce qu’on a discuté concernant les hypothèses du monde à ARN est relativement agnostique de cette notion.
Voir par exemple cette review
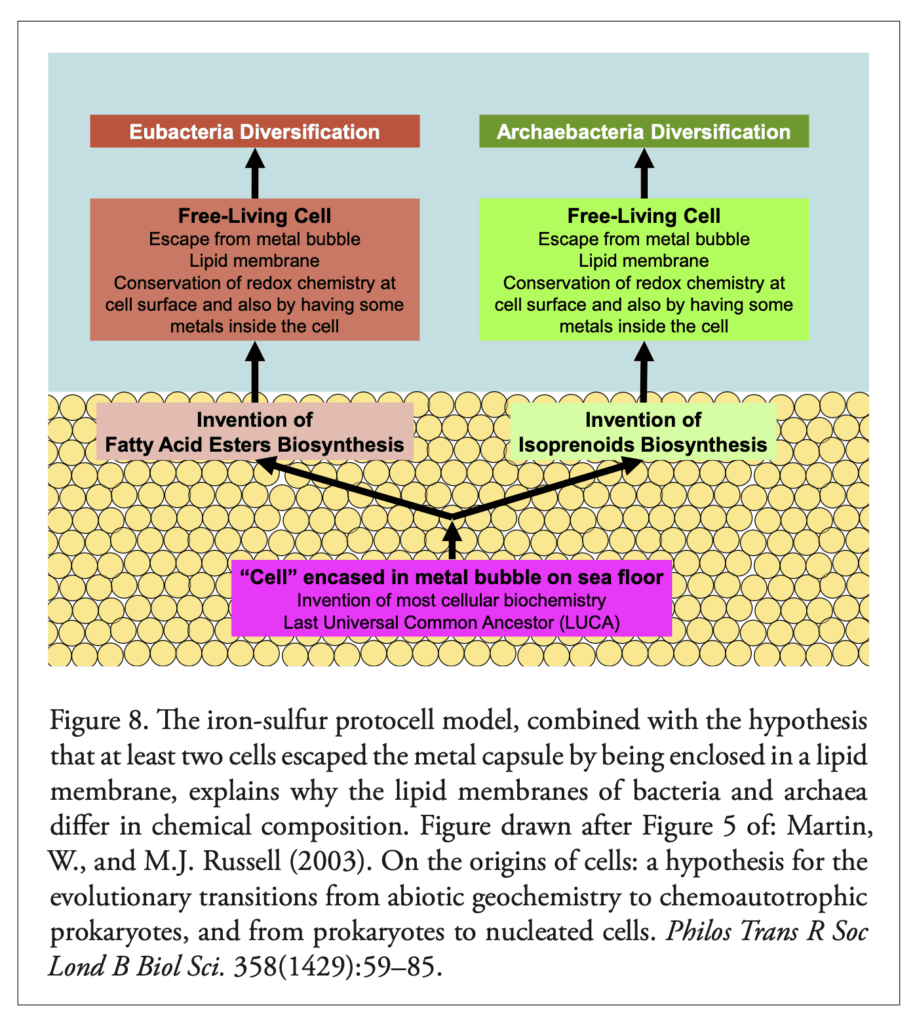
On pourrait imaginer une émergence du monde à ARN aussi bien dans des compartiments du type « proto-cellules », que dans des compartiments naturels comme certaines roches poreuses.
Voir par exemple cet article qui présente les termes de la discussion sous la forme d’un débat que l’on peut organiser entre des élèves
Bibliographie
Et pour finir, une petite bibliographie des papiers que j’ai lu pour préparer cette vidéo :
Ferris, J. P., Hill, A. R., Jr., Liu, R., & Orgel, L. E. (1996). Synthesis of long prebiotic oligomers on mineral surfaces. Nature, 381(6577), 59–61. https://www.nature.com/articles/381059a0
Johnston, W. K., Unrau, P. J., Lawrence, M. S., Glasner, M. E., & Bartel, D. P. (2001). RNA-catalyzed RNA polymerization: Accurate and general RNA-templated primer extension. Science, 292(5520), 1319–1325. https://doi.org/10.1126/science.1060786
Doudna, J. A., & Cech, T. R. (2002). The chemical repertoire of natural ribozymes. Nature, 418(6894), 222–228. https://doi.org/10.1038/418222a
Seelig, B. (2008). An autocatalytic network for ribozyme self-construction. Nature Chemical Biology, 4(11), 654–655. https://doi.org/10.1038/nchembio1108-654
Lincoln, T. A., & Joyce, G. F. (2009). Self-sustained replication of an RNA enzyme. Science, 323(5918), 1229–1232. https://doi.org/10.1126/science.1167856
Powner, M. W., Gerland, B., & Sutherland, J. D. (2009). Synthesis of activated pyrimidine ribonucleotides in prebiotically plausible conditions. Nature, 459(7244), 239–242. https://doi.org/10.1038/nature08013
Mulkidjanian, A. Y., Bychkov, A. Y., Dibrova, D. V., Galperin, M. Y., & Koonin, E. V. (2012). Origin of first cells at terrestrial, anoxic geothermal fields. Proceedings of the National Academy of Sciences, 109(14), E821–E830. https://doi.org/10.1073/pnas.1117774109
Robertson, M. P., & Joyce, G. F. (2012). The origins of the RNA world. Cold Spring Harbor Perspectives in Biology, 4(5), a003608. https://doi.org/10.1101/cshperspect.a003608
Cech, T. R. (2012). The RNA world in context. Cold Spring Harbor Perspectives in Biology, 4(7), a006742. https://doi.org/10.1101/cshperspect.a006742
Higgs, P. G., & Lehman, N. (2015). The RNA world: Molecular cooperation at the origins of life. Nature Reviews Genetics, 16(1), 7–17. https://doi.org/10.1038/nrg3841
Horning, D. P., & Joyce, G. F. (2016). Amplification of RNA by an RNA polymerase ribozyme. Proceedings of the National Academy of Sciences, 113(35), 9786–9791. https://doi.org/10.1073/pnas.1610103113
Meinert, C., Myrgorodska, I., de Marcellus, P., Buhse, T., Nahon, L., Hoffmann, S. V., … Meierhenrich, U. J. (2016). Ribose and related sugars from ultraviolet irradiation of interstellar ice analogs. Science, 352(6282), 208–212. https://doi.org/10.1126/science.aad8137
Usami, K., & Okamoto, A. (2017). Hydroxyapatite: Catalyst for a one-pot pentose formation. Organic & Biomolecular Chemistry, 15(42), 8888–8893. https://doi.org/10.1039/C7OB02051A
Kompanichenko, V. N. (2019). Exploring the Kamchatka geothermal region in the context of life’s beginning. Life, 9(2), 41. https://doi.org/10.3390/life9020041
Hud, N. V. (2019). RNA nucleosides built in one prebiotic pot. Science, 366(6461), 32–33. https://doi.org/10.1126/science.aaz1130
Becker, S., Feldmann, J., Wiedemann, S., Okamura, H., Schneider, C., Iwan, K., … Carell, T. (2019). Unified prebiotically plausible synthesis of pyrimidine and purine RNA ribonucleotides. Science, 366(6461), 76–82. https://doi.org/10.1126/science.aax2747
Damer, B., & Deamer, D. (2020). The hot spring hypothesis for an origin of life. Astrobiology, 20(4), 429–452. https://doi.org/10.1089/ast.2019.2045
Muchowska, K. B., Varma, S. J., & Moran, J. (2020). Non-enzymatic metabolic reactions and life’s origins. Chemical Reviews, 120(15), 7708–7744. https://doi.org/10.1021/acs.chemrev.0c00191
Wołos, A., Roszak, R., Żądło-Dobrowolska, A., Beker, W., Mikulak-Klucznik, B., Spólnik, G., … Grzybowski, B. A. (2020). Synthetic connectivity, emergence, and self-regeneration in the network of prebiotic chemistry. Science, 369(6511), eaaw1955. https://doi.org/10.1126/science.aaw1955
Blokhuis, A., Lacoste, D., & Nghe, P. (2020). Universal motifs and the diversity of autocatalytic systems. Proceedings of the National Academy of Sciences, 117(41), 25230–25236. https://doi.org/10.1073/pnas.2013527117
Yu, A. M., Gasper, P. M., Cheng, L., Lai, L. B., Kaur, S., Gopalan, V., Chen, A. A., & Lucks, J. B. (2021). Computationally reconstructing cotranscriptional RNA folding from experimental data reveals rearrangement of non-native folding intermediates. Molecular Cell, 81(4), 870–883.e10. https://doi.org/10.1016/j.molcel.2020.12.017
Pavlinova, P., Lambert, C. N., Malaterre, C., & Nghe, P. (2022). Abiogenesis through gradual evolution of autocatalysis into template-based replication. FEBS Letters, 597(3), 344–379. https://doi.org/10.1002/1873-3468.14507





4 Comments
Salut, il y a un petit soucis avec le css de ton blog : quand on sélectionne du texte celui-ci devient blanc sur fond blanc.
Bien vu, je pense c’est corrigé !
Pourriez-vous divulguer un lien pointant sur les encarts animes de fond montrant les simulations de protéines/enzymes en action ? Merci.
Il y a un ingrédient important dont tu n’as pas parlé, c’est le temps. L’apparition de la vie s’est déroulée sur des millions d’années et des réactions chimiques improbables finissent par arriver (de même que gagner au loto est quasiment improbable, mais finit par arriver si on joue très longtemps).