 En principe, notre ADN sait beaucoup de choses sur nous. Mais il n’est pas si simple de faire parler la quantité immense d’information qu’il contient. En 2008, des chercheurs ont réussi à montrer qu’il est possible de lire nos origines géographiques dans nos gènes, moyennant une méthode statistique adaptée : l’analyse en composantes principales.
En principe, notre ADN sait beaucoup de choses sur nous. Mais il n’est pas si simple de faire parler la quantité immense d’information qu’il contient. En 2008, des chercheurs ont réussi à montrer qu’il est possible de lire nos origines géographiques dans nos gènes, moyennant une méthode statistique adaptée : l’analyse en composantes principales.
A la pêche aux gènes
Pour faire parler nos gènes, il y a deux difficultés à surmonter : transcrire les informations contenues dans notre ADN en données numériques, puis trouver les bonnes méthodes statistiques qui permettent d’exploiter ces données.
Pour réaliser leur étude publiée dans Nature [1], John Novembre et ses collaborateurs sont partis d’une population de 3192 individus européens dont ils ont réalisé un génotypage. Mais qu’est-ce que cela signifie, génotyper ?
Il faut savoir que dans ce genre de situation, on ne regarde jamais le génome entier des individus. D’une part cela représenterait une quantité d’information énorme, d’autre part 99.9% de notre génome est absolument identique chez tous les individus. Il faut donc se concentrer sur les endroits susceptibles de présenter des différences d’une personne à une autre.
Le polymorphisme nucléotidique
 Pour génotyper les individus de leur étude, les auteurs ont donc regardé ce qu’on appelle les polymorphismes nucléotidiques (ou SNP en anglais pour single-nucleotid polymorphism). Il s’agit d’un type particulier de différences génétiques : des endroits bien précis de l’ADN où seule une base est susceptible de varier d’un individu à l’autre.
Pour génotyper les individus de leur étude, les auteurs ont donc regardé ce qu’on appelle les polymorphismes nucléotidiques (ou SNP en anglais pour single-nucleotid polymorphism). Il s’agit d’un type particulier de différences génétiques : des endroits bien précis de l’ADN où seule une base est susceptible de varier d’un individu à l’autre.
Par exemple imaginez une région de l’ADN qui chez tout être humain soit nécessairement l’une de ces deux séquences : soit AGTCGAGTCACA, soit AGTTGAGTCACA. Il s’agit d’un cas de polymorphisme nucléotidique, car dans cette séquence, seule la 4ème base est susceptible de différer d’un individu à un autre.
Pour leur étude, les auteurs ont utilisé une puce (comme celle-ci contre) permettant d’analyser simultanément 500 568 SNP chez un individu, et ce avec seulement 250 nanogrammes de son ADN !
A l’issue de cette collecte, chacun des 3192 individus de l’étude est donc décrit par 500 568 nombres, un pour chaque SNP qui a été analysé par la puce. On se retrouve avec un gros paquet de données qui est un immense tableau à 3192 lignes et 500 568 colonnes ! Pas facile à interpréter ! Et c’est là que rentrent en jeu les méthodes de statistique.
Dans leur papier, les auteurs ont utilisé une méthode simple mais puissante : l’analyse en composante principales. Cette méthode est particulièrement bien adaptée quand on se retrouve dans une situation où l’on cherche à faire parler des données avec un très grand nombre de variables. Voyons comment elle fonctionne sur un exemple simple.
Un graphique vaut mieux qu’un long tableau
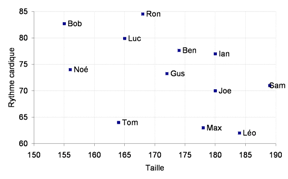 Imaginons un groupe de 12 lycéens dont on aurait mesuré la taille et la pulsation cardiaque au repos. On fabrique un tableau à 12 lignes et 2 colonnes, et on se demande comment on va faire parler ces données.
Imaginons un groupe de 12 lycéens dont on aurait mesuré la taille et la pulsation cardiaque au repos. On fabrique un tableau à 12 lignes et 2 colonnes, et on se demande comment on va faire parler ces données.
C’est un cas facile il n’y a que 2 variables : on n’a qu’à faire une représentation graphique !
On prend 2 axes, taille et rythme cardiaque, et chaque lycéen est représenté par un point. Le diagramme ci-contre montre le résultat dans notre cas fictif. Facile à lire, non ?
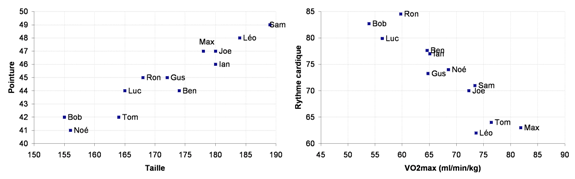
Alors compliquons un peu l’exercice : imaginons que vous ayez aussi mesuré la pointure de chaque individu, ainsi que sa VO2max (vous savez, le débit d’oxygène maximum qu’on peut atteindre pendant l’effort). Maintenant vous avez un tableau à 12 lignes et 4 colonnes. Sauf si vous savez voir en 4 dimensions, ça devient difficile à représenter graphiquement.
Une solution brutale, c’est de ne choisir que 2 variables parmi les 4 pour faire notre représentation. Voici deux possibilités ci-dessous : j’ai choisi à gauche la taille et la pointure, et à droite la pulsation et la VO2max.
Comme vous pouvez le constater, ces choix ne sont pas très judicieux car les points sont plus ou moins alignés. On s’en doutait : si on a la taille d’un individu, on connait en gros sa pointure. De même il existe un lien entre pulsation cardiaque et VO2max. Donc un graphique où les points sont alignés montre que les variables choisies sont redondantes. Tous les choix de variables ne se valent pas.
Même si on trouve 2 variables qui ne sont pas redondantes, le problème est qu’en n’en choisissant que 2 parmi toutes les colonnes dont on dispose, on a vraiment l’impression de jeter de l’information à la poubelle. C’est là que l’analyse en composantes principales vient à notre secours : c’est une méthode qui va chercher toute seule à fabriquer de nouveaux axes pertinents, en faisant un mélange de toutes les variables d’origine.
L’analyse en composante principales
 Essayons de trouver à la main des axes qui soient des combinaisons intelligentes : par exemple je peux choisir de créer un axe 1 qui serait une combinaison de la taille et de la pointure (par exemple pointure + 0.25*taille), et un axe 2 qui combine pulsation et VO2max (par exemple 1.5*pulsation-VO2max).
Essayons de trouver à la main des axes qui soient des combinaisons intelligentes : par exemple je peux choisir de créer un axe 1 qui serait une combinaison de la taille et de la pointure (par exemple pointure + 0.25*taille), et un axe 2 qui combine pulsation et VO2max (par exemple 1.5*pulsation-VO2max).
Voici à droite le résultat : j’ai une représentation graphique en 2 dimensions de mes données qui combine de manière intelligente mes 4 variables disponibles. Cette représentation disperse bien les points : les axes choisit ne sont pas redondants.
L’analyse en composante principale a pour objet de réaliser ce choix d’axes de manière « optimale ». Vous lui donnez votre tableau de données, et elle vous fabrique automatiquement deux axes qui sont des combinaisons de toutes les variables, et qui permettent de bien représenter les données en 2 dimensions.
 Maintenant imaginons que parmi mes lycéens, Max, Léo et Sam fassent partie de l’équipe de basket. Si on les colorie d’une couleur particulière sur le graphique, on repère tout de suite que se matérialise une zone précise contenant les joueurs de basket (en bas à droite).
Maintenant imaginons que parmi mes lycéens, Max, Léo et Sam fassent partie de l’équipe de basket. Si on les colorie d’une couleur particulière sur le graphique, on repère tout de suite que se matérialise une zone précise contenant les joueurs de basket (en bas à droite).
Votre graphique acquiert alors un pouvoir prédictif : si on vous donne les mesures d’un individu que vous ne connaissez pas, vous pouvez le placer dans le graphique : et s’il tombe en bas à droite, vous saurez qu’il y a une forte probabilité qu’il fasse partie de l’équipe de basket !
Bon sur cet exemple simpliste, on aurait pu le deviner. Mais voyons ce que ça donne sur le cas de données génétiques en grande quantité.
Et maintenant avec 500 000 variables
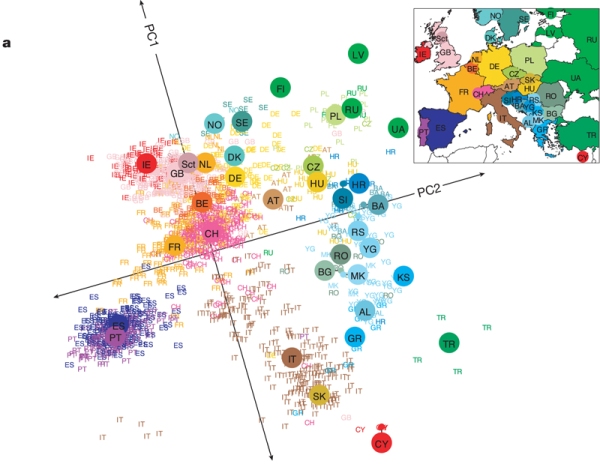
J. Novembre et ses collaborateurs ont d’abord réalisé un premier tri de nettoyage, qui leur a permis de se concentrer sur 1387 individus et environ 200 000 variables de SNP. Ils ont ensuite appliqué l’analyse en composantes principales. Cette dernière a donc fabriqué deux axes qui sont des combinaisons des 200 000 variables analysées. On peut les voir comme des coordonnées génétiques, qui résument en 2 dimensions le génome des individus.
Ils ont ensuite fait un graphique où chaque individu est représenté par un point dans ce nouveau système d’axe. Puis à chacun de ces points ils ont attribué une couleur symbolisant l’origine géographique de l’individu concerné. Et voici le résultat. La figure de gauche montre l’ensemble des individus dans le système d’axes fabriqué par l’analyse, et la carte à droite explique le code couleur.
Si vous regardez attentivement cette carte génétique et la position des différents individus, vous verrez qu’elle ressemble beaucoup à la carte de l’Europe. La cartographie en 2 dimensions de notre génome reproduit assez fidèlement la cartographie géographique de nos pays d’origine !
On constate plusieurs choses amusantes : les Alpes et les Pyrénées sont des barrières géographiques tout autant que génétiques : les Français sont significativement éloignés de leurs voisins espagnols et italiens. Un zoom sur la Suisse révèle que les 3 groupes linguistiques de ce pays sont même assez bien séparés. Il y a quand même quelques bizarreries : le seul individu slovaque de l’étude atterrit au milieu des Italiens.
A un niveau plus détaillé, les auteurs ont montré que l’axe Nord/Sud contenait plus de variabilité génétique que l’axe Est/Ouest. Cela semble avoir du sens du point de vue de la différenciation génétique en fonction du climat. On peut aussi imaginer que les migrations à latitude identique soient plus faciles.
Ils ont ensuite vérifié que les deux coordonnées génétiques constituent un très bon prédicteur de notre origine géographique. En clair ça marche comme dans mon exemple des joueurs de basket : vous analysez votre ADN avec une puce, vous en tirez les 200 000 variables SNP et en fonction de ces variables vous calculez votre position dans le diagramme : cette position va permettre avec une très bonne probabilité de déterminer votre origine géographique, sans la connaître a priori bien sûr !
Une telle méthode peut servir par exemple à identifier la provenance d’un criminel à partir de son ADN, mais on peut aussi imaginer plein d’applications plus ou moins réjouissantes de ce résultat.
Un grand merci à Benjamin qui m’a fait connaître cet article !
PS pour les spécialistes : je suis toujours épaté quand une méthode d’analyse non-supervisée comme l’ACP se révèle avoir un pouvoir prédictif comme si elle était supervisé. Chance ou truc profond ?
[1] John Novembre et al., Genes mirror geography within Europe, Nature 456,p98 (2008)
Sur les SNP : cours de génétique à l’UPMC




11 Comments
Très bien, j’ai lu en diagonale et je n’ai pas trouvé une seule erreur 🙂 Si ce n’est peut-être que l’article de J Novembre est un bon coup éditorial, mais que des ACP pour séparer les populations on en faisait depuis longtemps ! Je crois que c’est Lewontin qui a introduit cette technique en premier (sur des données beaucoup moins riches que les genome wide utilisés par J Novembre bien sûr).
Je peux ajouter un truc vite fait : c’est que les CP suivantes contiennent également beaucoup d’info qui ne peut pas s’interpréter aussi facilement par projection sur une carte 2D… c’est assez évident que la proximité géographique doit être bien corrélée avec les premières PC, tu prends n’importe quel modèle de diffusion de gènes du genre « stepping stones » (pas japonais), ça doit donner ça ; mais j’ai jamais vu de papier où on faisait le calcul (on s’y met ?).
Apparemment aussi c’est une bonne chose qu’il n’y ait pas trop de finlandais dans l’étude, j’ai vu récemment une carte factorielle où leur présence nombreuse déforme complètement la projection, transformant l’Europe dans une sorte de boomerang dont ils sont l’une des branches. Ces formes en boomerang (ou aile d’avion) sont classiques aussi quand on met Europe + Moyen Orient.
À partir des 15 microsatellites utilisés en « forensic », on ne peut pas être aussi précis mais on peut faire grosso modo Europe / Afrique / Asie, ce qui est complètement contraire à l’esprit de la loi sur les empreintes génétiques. Je peux te retrouver les réfs de ça si tu veux.
Je me suis régalé en lisnat cet article ^-^ 🙂
Pingback: 42: Guerre des sexes : la fin ? | Neuromonaco
Pingback: La drôle d’histoire de notre (in)tolérance au lactose | Science étonnante
Pingback: Les Sentinelles : le peuple le plus isolé du monde ? | Science étonnante
Encore une fois l’article est passionnant! L’ACP débouche forcement sur ces deux axes qui nous arrangent bien? Ou alors il y a d’autres jeux d’axes possibles qui serait « optimums »?
On pourrait prendre les autres composantes de l’ACP (3eme, 4eme, etc.) mais elles sont moins pertinentes (mathématiquement) que les 2 premières.
Elles ne sont pas « moins pertinentes » mais elles représente moins de variabilité sur l’ensemble de mes données. Si mon problème est de rechercher les mâles et les femelles je ne pense pas que les 2 ou 3 (en trois D ça se fait souvent) premières composantes me seront très utiles.
En général pour les données génétiques (en tout cas celles qui ne sont pas continues mais binaires du type présence ou absence d’un marqueur) on utilise l’Analyse Factorielle des Correspondances AFC basée sur des distances de khi-deux ( χ²).
« La cartographie en 2 dimensions de notre génome reproduit assez fidèlement la cartographie géographique de nos pays d’origine ! » dites vous.
Oui chez les animaux on appelle ça des races (exemple les éléphants d’Afrique et d’Asie qui ne sont pas deux espèces différentes mais deux races d’une même espèce.
Ne confondez pas « race » et « racisme » la race a un sens biologique et non le « racisme ». Le mot « race » a été rayé du vocabulaire français mais pas du tout de celui des anglo-saxons qui ne pense pas que l’on soigne la fièvre en cassant le thermomètre.
Homo Sapiens n’est pas « une race » mais une espèce au sens biologique du terme. Les « races » c’est ce que vous découvrez sur votre cartographie.
Note : je suis moi même un mélange de plusieurs « races » et je suis de plus généticien des populations.
Pan pour moi sur le bec ! l’exemple des éléphants n’est pas bon ! il s’agit de deux espèces. bon vous en trouverez un autre….les loups par exemple (sauf erreur de ma part)..cela n’enlève rien au reste de mes remarques.